Rozšíření možností genového inženýrství rostlin pomocí multiplexovatelných editačních systémů
5.7.2021 |
Vědci na univerzitě Martina Luthera v Halle-Wittenberg (MLU) a Leibnizově institutu rostlinné biochemie (IPB) použili vylepšené verze nástroje pro úpravy genů CRISPR/Cas9 k současnému vyřazení až dvanácti genů v rostlinách. Studie byla publikována v časopise The Plant Journal. Možnosti systému CRISPR/Cas pro editaci rostlinných genomů rozšířil také kolektiv amerických a čínských vědců. Pracovali se systémem CRISPR Cas12a, což je slibný systém se zajímavými vlastnostmi využitelný také pro multiplexování. Výsledky své práce publikovali v prestižním časopisu Nature communications.
Dědičnost rostlinných znaků není zpravidla tak jednoduchá a přímá, jak ji popsal Gregor Mendel, jehož experimenty týkající se dědičnosti u hrachu položily v 19. století základ genetiky. Mendel měl vlastně štěstí – u znaků, které studoval, platilo pravidlo, že danou vlastnost (např. barvu) určuje pouze jeden gen. Situace je ale mnohdy komplikovanější - často se na projevení určitého znaku podílí různé geny ve vzájemné interakci, nebo se může stejný znak projevit díky činnosti odlišných genů. V těchto případech inaktivace jednoho z těchto genů nevede k viditelnému účinku, což platí i u rostlin.
Dosud bylo vytváření více mutací u jedné rostliny složitým procesem - rostliny musely být např. upravovány po etapách pro každou jednotlivou mutaci a poté navzájem kříženy. Situaci by mohlo zjednodušit multiplexování, tedy cílení na dva nebo více lokusů pomocí společného (sdíleného) cílového místa nebo použití více programovatelných nukleáz k cílení na několik nezávislých míst. Vzhledem k relativní složitosti návrhu konstruktů existuje pouze několik příkladů, kdy byly pro multiplexování použity zinkové „prstové“ nukleázy (ZFN) nebo transkripčním aktivátorům podobné efektorové nukleázy (TALENs). Zaměření na více cílů se stalo mnohem snazší s objevem nukleáz řízených RNA (angl. RNA-guided nucleases, RGNs).
Ačkoli se přesné způsoby programování cílů liší, k nasměrování kterékoli z běžně používaných RGN (SpCas9, SaCas9 nebo Cas12) na více cílů postačí v zásadě „dodání“ nebo exprese dalších naváděcích RNA (gRNA). Pro editaci genomu rostlin jsou komponenty kódující systém RGN/gRNA nejčastěji stabilně integrovány do genomu rostlin pomocí transformace zprostředkované bakteriemi rodu Agrobacterium, kdy se pak nukleázový systém v rostlinných buňkách exprimuje.
Dosud byly sgRNA (single guide RNAs, jednoduché gRNA) v konstruktech s přenosovou DNA (T-DNA) pro multiplexování exprimovány jako jednotlivé transkripční jednotky (TU) pomocí promotorů přepisovaných RNA polymerázou III (Pol-III) (např. promotory U6 nebo U3) nebo jako polycistronní transkripty promotory promotorů přepisovaných RNA polymerázou II (Pol-II) (RNA polymeráza II) nebo Pol-III. Ve druhém případě primární transkript vyžaduje zpracování, kterého je dosaženo pomocí endogenního systému zpracování transferové RNA (tRNA), endonukleázy Csy4 nebo ribozymových sekvencí. Jednotlivá jednotka sgRNA měří 200-300 nukleotidů (nt) při použití promotorů Pol-III (v závislosti na regulačních prvcích) a může mít jen 130 nt při použití systému Csy4. Z „inženýrského hlediska“ je tedy integrace více jednotek sgRNA do transformačního konstruktu rostliny relativně jednoduchá.
V souladu s tím se RGN používají také v multiplexním režimu a k dispozici jsou sady nástrojů pro sestavení příslušných komplexních nukleázy kódujících konstruktů. Při použití většího počtu sgRNA pro multiplexování však lze očekávat sníženou účinnost (soutěž o společné „zdroje“), umlčení exprese transgenu nebo rekombinace. To jsou pravděpodobně důvody, proč je relativně málo příkladů, kdy byl pro multiplexování použit zvýšený počet sgRNA (≥6). Celkově zatím zůstává nejasné, kolik cílových míst lze multiplexováním řešit současně a efektivně.
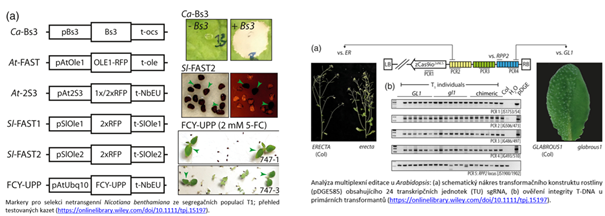
Němečtí vědci v jejich studii zkoumali účinnost a limity multiplexní editace u dvou hlavních modelových druhů dvouděložných rostlin - Arabidopsis thaliana a Nicotiana benthamiana. Nejprve rozšířili stávající sadu nástrojů pro sestavování konstruktů s až 32 sgRNA a vyladili používanou sadu nástrojů pro lepší účinnost začleněním vysoce intronově optimalizované varianty genu Cas9, zCas9i. Pro multiplexování v N. benthamiana vyvinuli nové markery pro selekci transgenů. Zacílili na osm genů pomocí sady devíti různých sgRNA. U A. thaliana zacílili 12 různých genů pomocí 24 sgRNA. Ukázali, že multiplexováním u N. benthamiana lze dosáhnout velmi vysoké účinnosti multiplexování. Identifikovali bialelické mutace téměř na všech cílových místech u náhodně vybraných netransgenních jedinců T1 a pozorovali neočekávané nadměrné zastoupení homozygotních mutací. Při editaci 12 genů A. thaliana pomocí 24 sgRNA izolovali také linii s 12 mutacemi bez transgenů; současně však pozorovali nižší účinnost. Výsledky ukazují, že limitujícím faktorem při aplikacích multiplexování vyššího řádu se stává dostupnost Cas9, zatímco rekombinace a umlčování transgenů se nejeví jako problematické. Autory uváděné vysoké účinnosti multiplexní editace otevírají nové perspektivy pro generování komplexních genotypů a pro funkční analýzu mnoha kandidátních genů pomocí RGN.
Druhý typ nukleázy, který zmiňujeme v úvodu, nukleáza Cas12a (dříve známá jako Cpf1), vykazuje několik klíčových rozdílů oproti Cas9:
- Při štěpení vytváří "lepivé" konce ve dvouvláknové DNA na rozdíl od "tupých" konců vytvářených Cas9. Lepivé konce napomáhají začlenění nových sekvencí DNA, takže Cas12a je při zavádění genů účinnější než Cas9.
- Cas12a vyžaduje PAM sekvenci (krátká sekvence sousedící s oblastí DNA určenou ke štěpení, která je nezbytná pro štěpení Cas nukleázou) bohatou na thymin (tím poskytuje alternativní cílová místa k Cas9) a k úspěšnému cílení vyžaduje pouze crRNA (Cas9 vyžaduje navíc i trans-aktivační crRNA, tracrRNA). Malé crRNA Cas12a se tedy jeví jako vhodnější pro multiplexní editaci genomu, protože jich lze do jednoho vektoru zabalit více než sgRNA Cas9.
- Cas12a dále štěpí DNA 18-23 párů bází za místem PAM. Po opravě tak nedochází k narušení rozpoznávací sekvence a může proběhnout více kol štěpení DNA. To by teoreticky mělo zvýšit úspěšnost editace genomu. Naproti tomu, protože Cas9 stříhá pouze 3 páry bází před místem PAM, dochází cestou nehomologního spojování konců (angl. non-homologous end joining, NHEJ) k indelovým mutacím, které ničí rozpoznávací sekvenci, a tím brání dalším kolům štěpení.
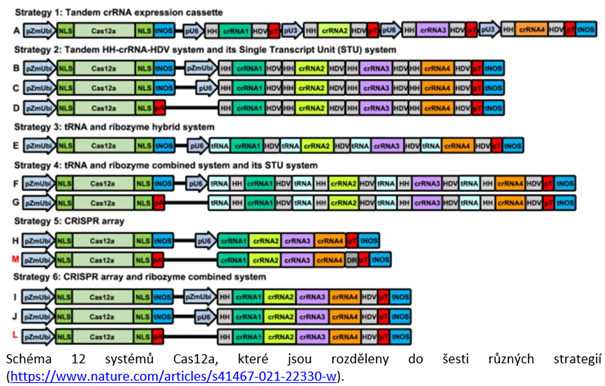
Aby rozšířili možnost použití Cas12a, prověřili američtí a čínští vědci devět ortologů Cas12a, které se nevyskytují u rostlin a zjistili, že šest z nich (ErCas12a, Lb5Cas12a, BsCas12a, Mb2Cas12a, TsCas12a a MbCas12a) vykazuje vysokou editační aktivitu v rostlinách rýže. Mezi nimi vyniká Mb2Cas12a s vysokou editační účinností a tolerancí k nízké teplotě. Upravená varianta Mb2Cas12a-RVRR umožňuje editovat rýži s mírnějšími požadavky na PAM a poskytuje dvakrát větší pokrytí genomu než divoký typ SpCas9. Autoři také porovnali 12 multiplex systémů s Cas12a a identifikovali účinný systém, který vykazuje téměř 100% účinnost bialelické editace se schopností cílit v rýži až na 16 míst. Jedná se o dosud nejvyšší úroveň multiplexních editací u rostlin pomocí Cas12a. Dále vědci vyvinuli dva kompaktní interferenční systémy CRISPR-Cas12a s jednou transkripční jednotkou pro současné potlačení více genů v rýži a A. thaliana. Publikovaná studie tak výrazně rozšiřuje rozsah použití Cas12a pro genové inženýrství plodin.
Zdroje:
- https://onlinelibrary.wiley.com/doi/10.1111/tpj.15197
- https://www.nature.com/articles/s41467-021-22330-w
- https://www.sciencedirect.com/science/article/pii/S2319417019305050